报告人:中国科学院北京生命科学研究院 赵方庆教授 记录人:李婷 肖淳夫(李川昀实验室)
2021年11月2日下午,受北京大学未来技术学院分子医学研究所汪阳明教授邀请,中国科学院北京生命科学研究院赵方庆教授在未来技术学院报告厅带来了一场题目为“生物大数据挖掘技术”的精彩报告。
背景
生物大数据的有效挖掘和利用一直以来是生命科学和医学研究的关键环节。随着多组学技术的广泛应用和大数据的指数级增长,也带来了一些问题,例如:数据规模往往十分庞大,且具有高维、多噪、异质等属性。从数据到信息到知识,我们需要一系列方法的开发和应用来解读生物学大数据。赵方庆实验室针对过去几年涌现出的组学数据,建立了高效的统计学模型和算法工具,解析其中的功能以及与疾病的关联。
研究方向1 环形RNA
从上世纪70年代人们看见环形RNA分子开始到2013年Nature论文发现环形RNA可能具有一些功能,环形RNA逐渐引起了研究者的广泛关注。环形RNA的种类很多,但是表达丰度很低,只占转录本数量的0.1-2%,能够区分出环形的概率更低(10-20%),所以很少能检测出来;另外,检测的信噪比很低,99%的都是噪音。基于此,近几年,赵方庆团队在环形RNA的识别,重建(定量),以及功能进行了一系列的探索,也取得了非常多的研究成果。
环形RNA识别的质控算法
由于真核生物转录和剪接的复杂性以及不同来源环形RNA的差异表达,已有的几种检测环形RNA的算法存在低敏感度、低可靠性、运行时间长、内存使用率高以及管道复杂等缺陷。因此,赵方庆团队建立了基于多重种子匹配策略的环形RNA质控方法,显著降低了假阳性率。
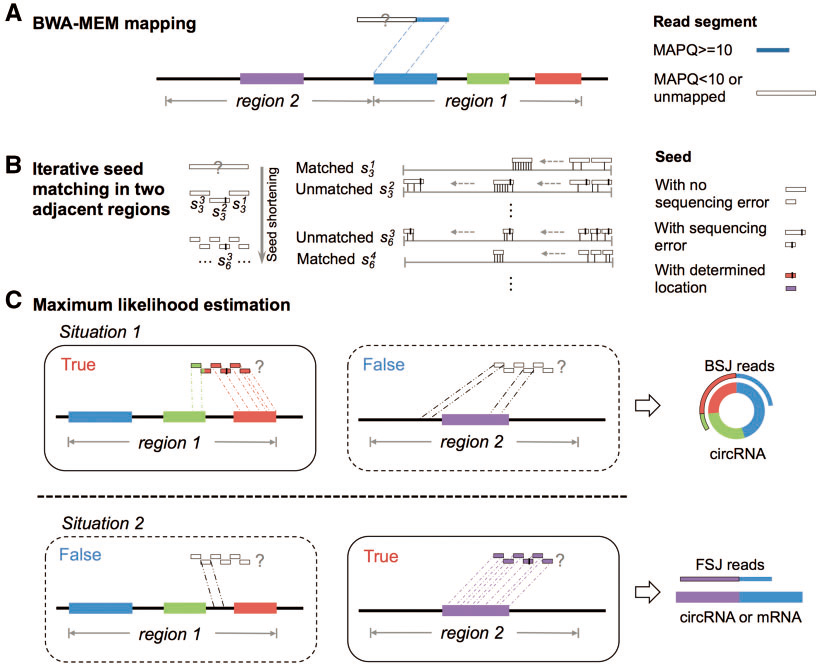
Figure 1 The MLE-based algorithm implemented in CIRI2. (Gao et al., 2018)
环形RNA内部结构重建和可变剪接识别
由于尚没有研究对环形RNA的内部结构进行探索,因此,赵方庆团队开发了新的环形RNA外显子检测算法CIRI-AS,通过结合长读长测序数据以及实验验证,全面研究了10个人类细胞系和62个果蝇样本中环状RNA的内部结构,发现可变剪接普遍存在,并且解释了其剪接模式与mRNA可变剪接的显著不同。

Figure 2 The workflow of cirexon and AS detection. (Gao et al., 2016)
环形RNA转录本重建和定量
目前,环形RNA的研究正从环状转录本的鉴定转向其生物学功能的探索。然而,这些研究受到了其全长序列的大规模鉴定以及无法在亚型水平上进行准确定量的限制。因此,赵方庆团队首次提出环形RNA识别的新特征(reverse overlap),建立了环形RNA全长转录本重建的新方法,可以实现80%左右的环形RNA的全长重建。此外,也首次提出基于转录本水平的环形RNA定量方法,为准确挖掘差异环形RNA分子奠定了基础。
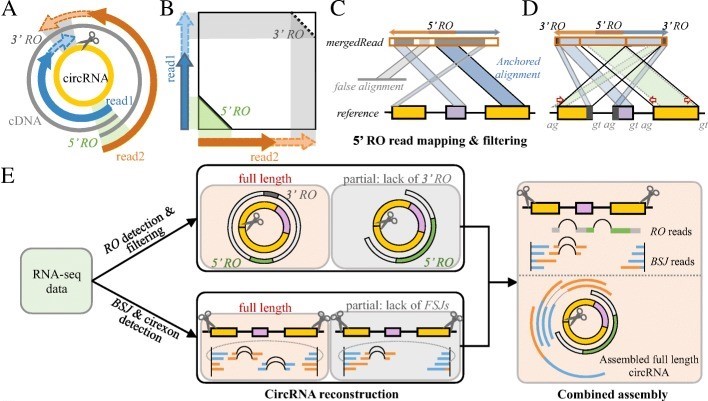
Figure 3 Workflow of reverse overlap detection and full-length circular RNA reconstruction. (Zheng et al., 2019)
环形RNA转录本精确定量
环形RNA的检测和定量面临几个重大挑战,包括假阳性率高、RNase R处理效率不稳定,以及对反向拼接连接读段的低估。不同的实验技术手段、不同物种以及批次效应都会给环形RNA的定量带来很大的偏差,因此定量化和标准化是一种极大的需求。环形RNA的定量对后续的功能研究和筛选也是必不可少的。因此,赵方庆团队提出了环形RNA无偏定量和差异表达分析的新方法(CIRIquant),并发现了两类剪接体转换事件:线性/环形成环比变换、成环位点偏好转换。
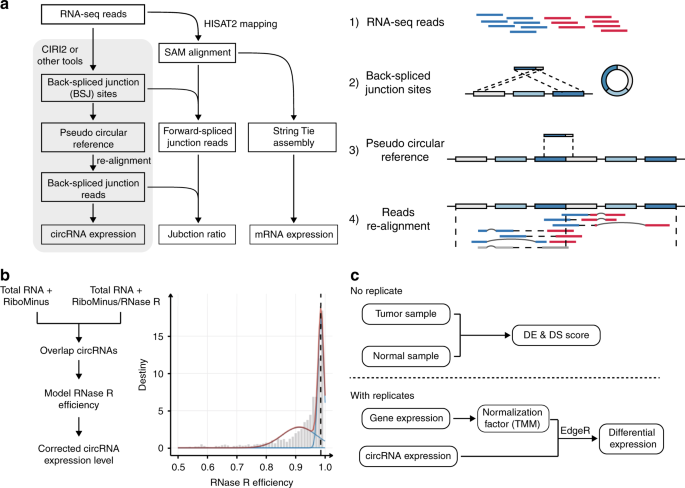
Figure 4 Workflow of circRNA quantification. (Zhang et al., 2020)
环形RNA的功能注释和挖掘
环状RNA是多种生物和疾病过程的重要调控因子,那么,如何从海量环形RNA分子中高效筛选出具有潜在生物学功能的分子?借鉴mRNA功能研究的方法,我们很容易想到从母本基因着手来研究环形RNA的功能,但是环形RNA与母本基因的功能并不相关。因此,为了全面了解环形RNA的多样性,赵方庆团队开展了人猴鼠多种组织的环形RNA库的大规模研究,建立了跨物种、多类型网络整合和注释方法,为环形RNA的功能筛选和注释奠定了方法学基础。
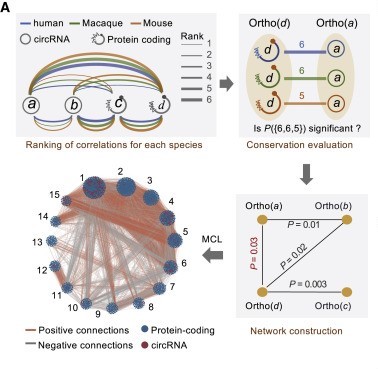
Figure 5 Construction of evolutionarily conserved co-expression network in human, macaque, and mouse. (Ji et al., 2019)
环形RNA的保守性评估和功能挖掘
现有的环形RNA数据库对转录组学的研究尤为重要。然而,大多数数据库都不适于挖掘深入的信息来确定候选环形RNA的优先级。为此,赵方庆团队提出保守环形RNA识别及保守性打分方法,建立跨物种多组织的环形RNA功能挖掘平台。他们发现,保守环形RNA倾向于有更高表达量、更保守表达模式、更高成环比以及更保守的剪接模式。
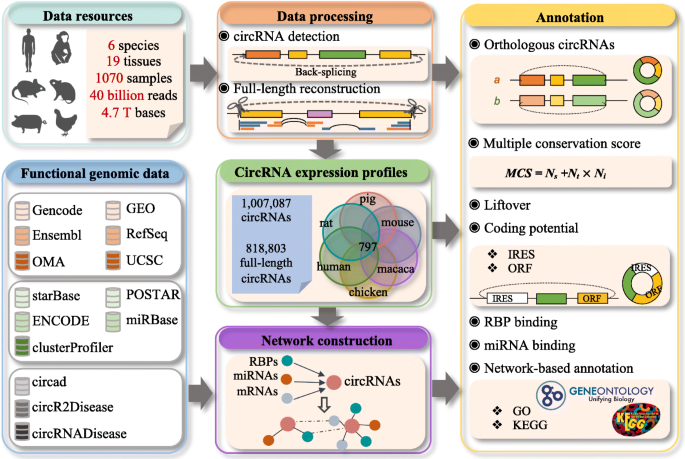
Figure 6 Schematic overview of the construction of circAtlas and associated functionalities. (Wu et al., 2020)
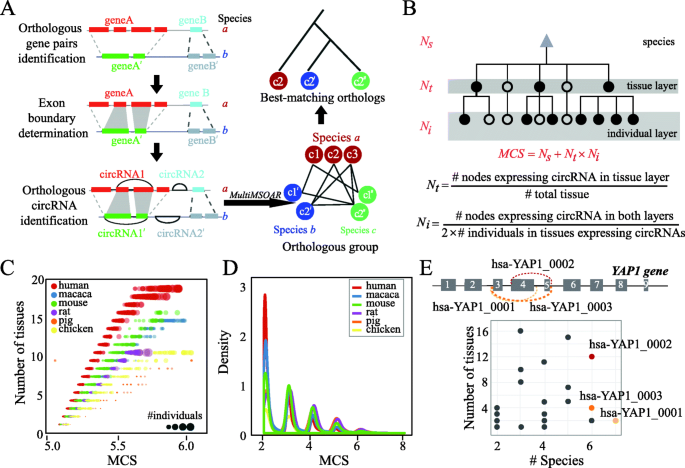
Figure 7 Workflow of the conserved circRNA identification. (Wu et al., 2020)
发现一类内含子自连型的环形RNA
在最近的工作中,赵方庆团队开发了一种使用纳米孔技术富集和全长测序环形RNA亚型的方案。相比之前的方法,实现了20倍高的富集。据此,发现了一种新型的内含子自连型的环形RNA,具有特殊的剪接和表达模式,并且这种环形RNA在进化上有强的保留性,说明有一定的功能。
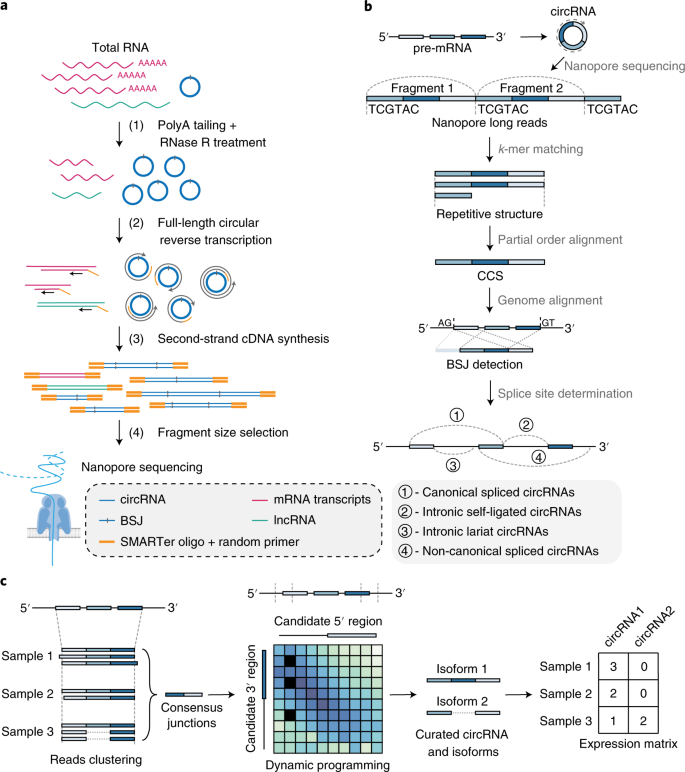
Figure 8 Method overview. (Zhang et al., 2021)
小结
赵方庆团队的工作完善了环形RNA的计算方法学,同时也带来了一些生物学的新发现。首先,他们建立了不依赖先验注释的环形RNA识别算法,并且首次提出环形RNA识别新特征及转录本重建算法,另外,提出环形RNA无偏定量和差异表达分析的新方法,也建立了基于多元网络的功能注释方法。基于这些方法,他们发现了新类型的intronic self-ligated circRNAs以及两类剪接体转换事件,并筛选获得一类高度保守的环形RNA(OO-type circRNA),也揭示环形RNA受到独立于线性转录本的调控作用。
研究方向2 微生物组结构及功能的精准解析技术
为什么要研究微生物组?从数据角度:菌群的物种复杂度高,不同物种的丰度不一样,因此菌群数据与RNA的数据有一定的相似性,在算法和数据挖掘方法上也有先天的相似性;从科学问题上而言,菌群在生活中无处不在,与人类的健康和疾病密切相关,特别是慢性病和复杂疾病,但是解析微生物与疾病的因果关系需要很高的精准度;如何解析菌群结构组成,如何挖掘菌群功能基因,如何追溯菌群动态变化;针对这些问题我们实验室建立了一系列实验技术和基本方法。
1、复杂微生物群落基因组结构解析技术
赵方庆团队首次提出菌群复杂度动态调控策略的组装算法,实现对复杂菌群基因组结构的准确解析;这种方法是一种基于流式细胞术和单细胞测序的分类迷你宏基因组方法,通过原始宏基因组的互补,能够高效地从分类后的迷你宏基因组中恢复高质量的基因组。此外,他们将该方法应用于一个未开发的海藻表面定殖菌群,一次成功地恢复了75个高质量的基因组。这种方法具有高效率、高精度等特点,将极大地改善从复杂或新的群落中获取微生物基因组的途径。目前该方法已应用于:中国微生物组计划预研项目、水圈微生物组重大研究计划、美国人体微生物组计划以及新加坡启动的污水微生物组计划等。
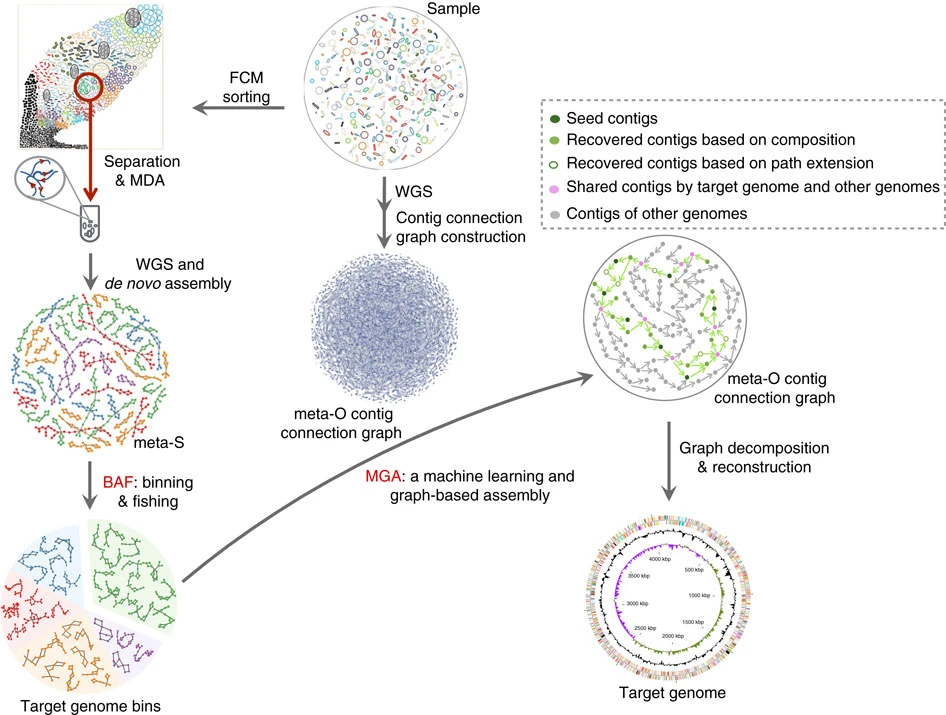
Figure 1. Overview of the metaSort approach. (Ji et al. 2017)
2、功能基因及其变异的靶向重建技术
赵方庆团队首次提出基于密码子的德布罗意图算法,用于编码基因的靶向重建,解决了碎片化和高冗余度现象;大多数基因预测方法是在没有密切相关的参考基因组的情况下从转录组组装中检测编码序列。由于高转录片段和广泛的装配错误,这些方法的应用有限,可能导致冗余或错误的编码序列预测。他们提出了inGAP-CDG,它可以从未组装的转录组中靶向性地构建全长和非冗余编码序列,使用基于密码子的de Bruijn图来简化组装过程,并使用基于机器学习的方法来过滤假阳性。与其他方法相比,inGAP-CDG的预测编码序列长度显著增加,对测序错误和不同读取长度的稳健性也显著提高。
Figure 2. Comparison between the traditional de Bruijn graph and the codon-based de Bruijn graph. (Peng et al. 2016)
3、菌群动态演替过程的追溯技术
(1)时间动态——口腔菌群演替:赵方庆团队解析了洁牙前后口腔菌群的动态变化,揭示微生物被膜从崩解到重建的演替规律;他们的研究揭示了强干扰后口腔微生物群的恢复和长期稳定性,并确定了最剧烈的群落变化和结构恢复的关键时间点和阶段。这些发现提示,当考虑使用口腔细菌作为生物标志物来预测消化系统疾病时,应考虑收集时间和地点。
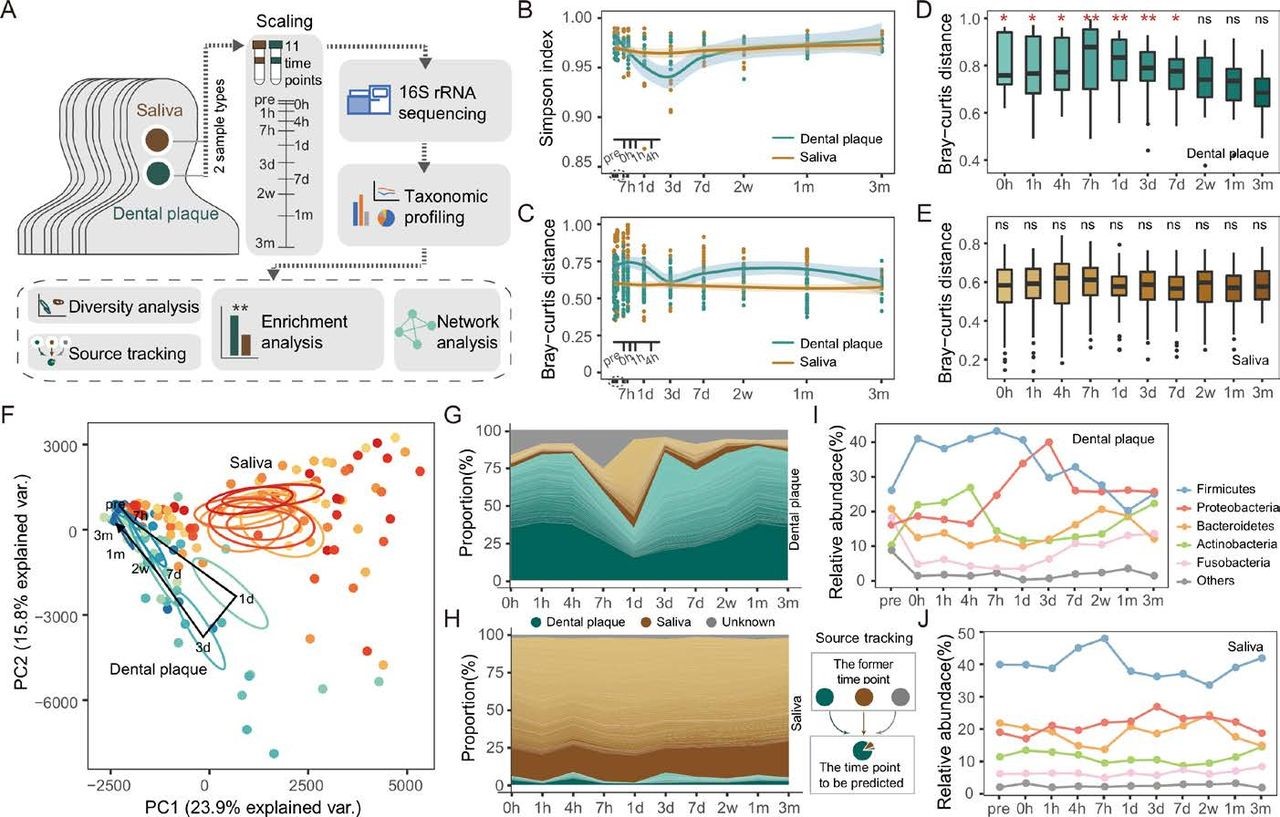
figure 3. Longitudinal dynamics of in vivo human oral microbiota. (Wang et al. 2020)
(2)空间动态——母婴菌群传递:赵方庆团队首次发现妊娠期糖尿病对孕妇、新生儿菌群的趋同塑造,揭示母婴之间菌群交互对健康的影响和另一种形式的遗传,并强调了了解早期微生物群形成的重要性。
(3)与宿主交互动态——病原菌群传播:赵方庆团队等人对6种硬蜱进行了高质量基因组测序和组装,并对678份蜱标本进行了重新测序,以了解蜱的遗传多样性、种群结构和病原体分布三个关键方面,首次揭示了不同蜱种的遗传结构和病原体组成主要受生态和地理因素的影响;他们进一步确定了与不同寄主范围、生命周期和分布相关的物种特异性决定因素;这项研究揭示了宿主-媒介-菌群的交互关系,发现宿主类型与生态位对蜱传播病原体的决定性影响。
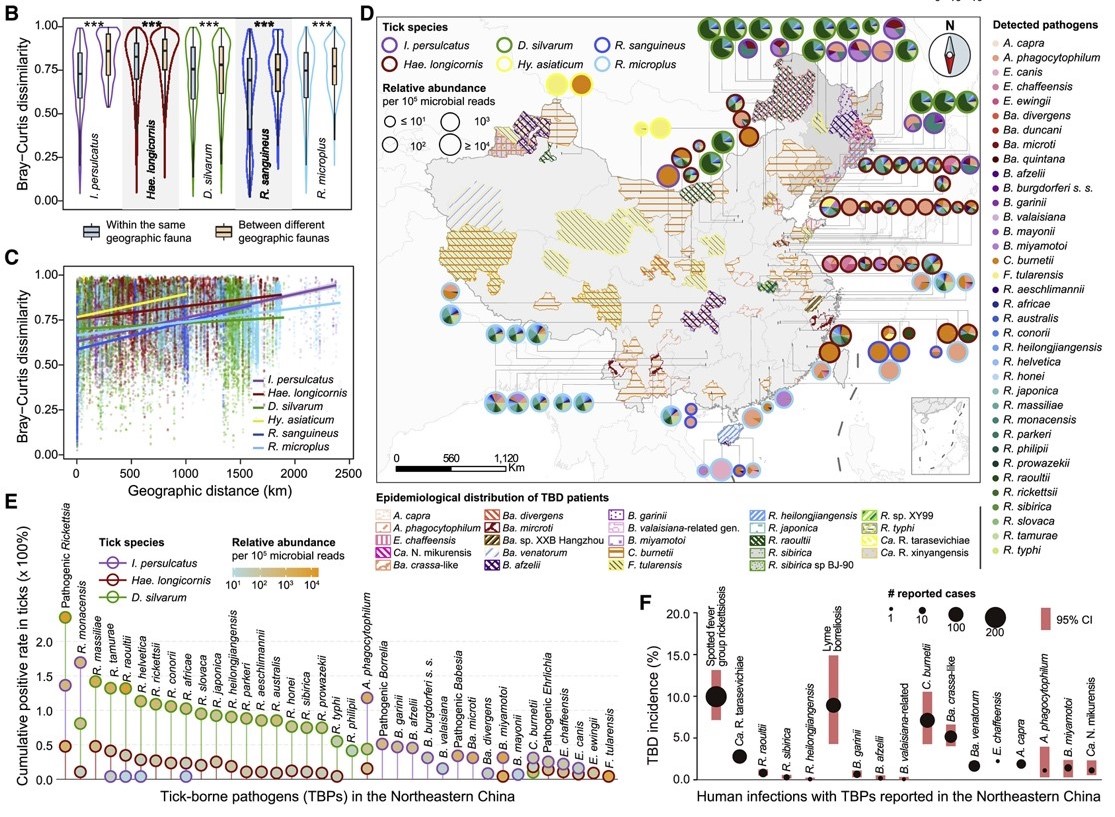
Figure 4. Potential Pathogen Profiling of Six Tick Species. (Jia and Wang et al. 2020)
(4)菌群异位与生殖健康——赵方庆团队分析了145名女性的阴道和子宫微生物样本,并结合深入挖掘的公共数据和动物实验来描述女性生殖道中的微生物易位及其在调节子宫健康中的作用。研究显示,随着年龄的增长,子宫和阴道微生物群发生同步变化和逐渐收敛。他们还发现,将某些阴道细菌移植到大鼠阴道内可诱导或减轻子宫内膜炎样症状,并证实了某些阴道细菌对子宫内膜的损伤或保护作用。本研究阐明了阴道细菌易位与子宫微生态和子宫内膜健康的相互依赖关系,揭示了上下生殖道菌群的动态交互。
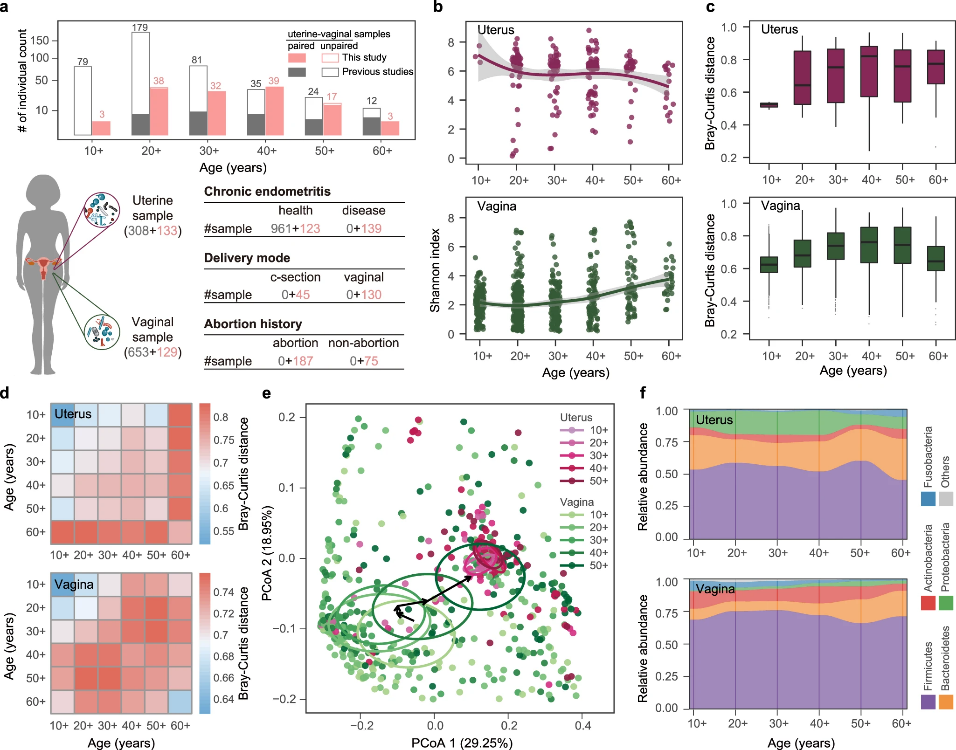
Figure 5. Cohort overview and variations in uterine and vaginal microbiome associated with aging. (Wang, Li and Ma et al. 2021)
小结
赵方庆团队的工作针对微生物组建立了一系列实验技术和基本方法,实现了对复杂菌群基因组结构的准确解析,构建了功能基因及其变异的靶向重建技术,实现了对菌群在时空动态演替过程、与宿主交互和异位的追溯,这些工作为微生物组学领域提供了高效工具和有力参考。
Q&A
在这次的精彩报告之后,赵方庆教授和在场师生进行了热烈的交流:
Q1、请问您每开发一个算法,是否会借鉴或者改进其他算法,怎么构思一个算法?
这也是我们带学生普遍面临的问题,目前做算法的人有两类:第一类主要是生物学背景的,根据需求来,例如为了分析环形RNA数据而开发方法;第二类就是统计系计算机系背景的人,他们数理背景很好,为了模型评价指标的优化来开发或改进模型;但实际上在生物领域中来说开发方法不应该先去套模型,应该看数据中有什么问题,根据需求导向;
Q2、如何评价针对环形RNA的富集建库方法?
现在不需要做富集,富集面临的问题有:首先要求高质量完整的环形RNA,一旦RNA消化后会影响真实的表达量;第二,一个基因既有线性也有环形,这两个结构之间动态的变化更能说明问题,并且现在测序很便宜,我不建议现在做富集,如果在之前确实可以节约成本。
Q3、如果发现一个新的环形RNA,需要做什么实验来鉴定,鉴定一个新的环形RNA的指标是什么?
首先是新的含义:第一个是之前没有报道过,但这样其实没什么意义,随便测一次就能发现很多新的环形RNA、lncRNA和miRNA,需要很谨慎,大部分都不太重要;但如果发现了新的类型,例如在线粒体中形成的环形RNA,或者如果找到一些特殊,例如表达量高、在不同条件下表达量差别很大、有新的机制,这种就值得继续去研究;
Q4、如果单细胞里面环形RNA组学有差异,其蛋白组表达是否有差异?
不一定,有些lncRNA和环形RNA有编码的潜力,它们的EST序列或者m6A修饰能够招募并启动翻译,但不能对整体蛋白表达有影响,主要是一些微调;
Q5、有些报道说大多数环形RNA是有害的?如何看待这些工作?
实验、进化和生物信息对现象的描述语言不一样,他们说的“有害”只是进化意义上的有害,在进化上如果不产生环形RNA是更保守的,但是它们在分子功能上不一定有害,这是两个层次的概念;其次从保守性来说,进化上的“有害”也只是总体上“有害”,少部分环形RNA事件确实有功能,也有很多实验证实,不能笼统的将所有环形RNA套进化“有害”的概念;
Q6、环形RNA的定位在细胞核里面多还是细胞质里面多?
有些环形RNA不出核,有些环形RNA出核,不同环形RNA偏好的差异性很大,之前有人研究过环形RNA的长度或者修饰例如m6A是否对出核有影响,但目前没有特别的规律;
Q7、有人在微生物中发现过环形RNA吗?
微生物中存在成环的RNA,但与正常的环形RNA完全不一样,比如一些植物病毒,它们的长度很长,但产生机制与exon形成的环形RNA完全不一样,tRNA上也有一小段的环形RNA,产生机制也与正常环形RNA不一样,我们的研究中从来没有发现微生物中存在真正的环形RNA,大部分都是假阳性;
参考文献
Gao, Y., Zhang, J., & Zhao, F. (2018). Circular RNA identification based on multiple seed matching. Briefings in Bioinformatics , 19 (5), 803–810. https://doi.org/10.1093/bib/bbx014
Gao, Y., Wang, J., Zheng, Y., Zhang, J., Chen, S., & Zhao, F. (2016). Comprehensive identification of internal structure and alternative splicing events in circular RNAs. Nature Communications , 7 (1), 12060. https://doi.org/10.1038/ncomms12060
Zheng, Y., Ji, P., Chen, S., Hou, L., & Zhao, F. (2019). Reconstruction of full-length circular RNAs enables isoform-level quantification. Genome Medicine , 11 , 2. https://doi.org/10.1186/s13073-019-0614-1
Zhang, J., Chen, S., Yang, J., & Zhao, F. (2020). Accurate quantification of circular RNAs identifies extensive circular isoform switching events. Nature Communications , 11 (1), 90. https://doi.org/10.1038/s41467-019-13840-9
Ji, P., Wu, W., Chen, S., Zheng, Y., Zhou, L., Zhang, J., Cheng, H., Yan, J., Zhang, S., Yang, P., & Zhao, F. (2019). Expanded Expression Landscape and Prioritization of Circular RNAs in Mammals. Cell Reports , 26 (12), 3444-3460.e5. https://doi.org/10.1016/j.celrep.2019.02.078
Wu, W., Ji, P., & Zhao, F. (2020). CircAtlas: An integrated resource of one million highly accurate circular RNAs from 1070 vertebrate transcriptomes. Genome Biology , 21 (1), 101. https://doi.org/10.1186/s13059-020-02018-y
Zhang, J., Hou, L., Zuo, Z., Ji, P., Zhang, X., Xue, Y., & Zhao, F. (2021). Comprehensive profiling of circular RNAs with nanopore sequencing and CIRI-long. Nature Biotechnology , 39 (7), 836–845. https://doi.org/10.1038/s41587-021-00842-6
Ji, P., Zhang, Y., Wang, J., & Zhao, F. (2017). MetaSort untangles metagenome assembly by reducing microbial community complexity. Nature communications, 8, 14306.
Peng, G., Ji, P., & Zhao, F. (2016). A novel codon-based de Bruijn graph algorithm for gene construction from unassembled transcriptomes. Genome biology, 17(1), 232.
Wang, J., Jia, Z., Zhang, B., Peng, L., & Zhao, F. (2020). Tracing the accumulation of in vivo human oral microbiota elucidates microbial community dynamics at the gateway to the GI tract. Gut, 69(7), 1355–1356.
Jia, N., Wang, J., Shi, W., et al. (2020). Large-Scale Comparative Analyses of Tick Genomes Elucidate Their Genetic Diversity and Vector Capacities. Cell, 182(5), 1328–1340.e13.
Wang, J., Li, Z., Ma, X., et al. (2021). Translocation of vaginal microbiota is involved in impairment and protection of uterine health. Nature communications, 12(1), 4191. https://doi.org/10.1038/s41467-021-24516-8
