汪阳明实验室最近鉴定了多个来源于微生物中的双链DNA脱氨酶,并将其中一种改造成为线粒体碱基编辑器(图1)。该项成果1于2023年2月16日在《自然-通讯》( Nature Communications )在线发表,论文题目为“DddA homolog search and engineering expand mitochondrial base editing”,论文链接https://www.nature.com/articles/s41467-023-36600-2。
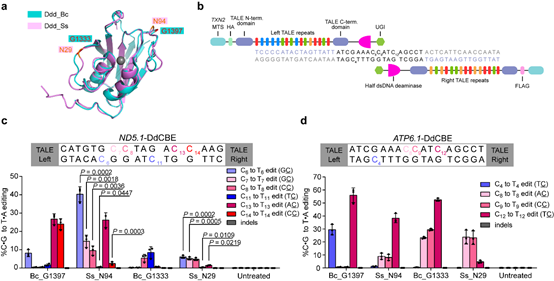
图1.思邈菌DddA衍生的线粒体碱基编辑器成功编辑多个线粒体基因组位点。
细胞中的生物学反应在种类繁多的细胞器中发生,其中线粒体十分特别,它是细胞的能量工厂,为细胞乃至人体活动提供能量,并且在多个层面参与细胞信号通路调控细胞的生死存亡和命运。更有意思的是,线粒体拥有自己的DNA。细胞中已知只有两种细胞器拥有自己的DNA,另一种是叶绿体。目前已发现有上百种线粒体DNA的突变能够导致人类疾病,包括耳聋、心血管疾病和神经退行性疾病。如何纠正这些DNA突变来治疗人类疾病,以及如何诱导特定的突变来模拟人类疾病和研究线粒体基因的功能,始终是领域内的难题。对于细胞核中的DNA,科学家们可以通过CRISPR系统结合DNA脱氨酶实现特定碱基的高效突变。然而该系统由于外源RNA难以高效转运至线粒体内部(有争议),在线粒体编辑中尚无实际应用。这一问题的解决得益于一类新的DNA脱氨酶的发现。与CRISPR系统联用的碱基编辑酶都是单链DNA脱氨酶,2020年左右Mougous实验室在伯克霍尔德菌中发现了一种脱氨酶,可以在双链DNA上工作,因此命名为双链DNA脱氨酶(Double strand DNA deaminase, DddA)。双链脱氨酶是细菌对抗不同种属细菌的一项利器,通过一种类似于注射器的装置,伯克霍尔德菌将双链脱氨酶注射进入其他细菌胞体内,造成基因组的广泛突变,从而杀死其他细菌。与David Liu实验室合作,他们将DddA与另一类基因编辑常用的蛋白TALE相结合,成功实现了线粒体DNA的碱基突变2。然而,这类碱基编辑器的序列兼容性较低,例如最初的碱基编辑器DdCBE只对T之后的C有较高的编辑效率,编辑后将C突变为U,该碱基将被识别为T。为了解决序列兼容性低的问题,领域内常用的方法有两种,一种是工程化改造原有编辑器,另一种是寻找其他种类的碱基编辑器。David Liu等人通过实验室进化的方法得到了可以编辑A, T和C之后的C的碱基编辑器3,但能够编辑G之后C的线粒体碱基编辑器仍然缺乏。
汪阳明实验室米黎同学在研究伯克霍尔德菌DddA的序列和结构时,发现其羧基端存在一段序列,这些序列对于DddA的脱氨活性十分重要(图2a)。利用这一重要信息,结合PSI-BLAST生物信息学分析手段, 研究者在现有微生物的基因组序列中鉴定了多个与DddA同源的双链脱氨酶。进一步的细致分析发现,来源于思邈菌( Simiaoa Sunni ; 以中国唐代“药王”孙思邈命名)的DddA同源基因(Ddd_Ss),具有能够编辑A,T和G之后C的活性(图2b),从而有可能弥补目前线粒体碱基编辑器的缺口。作者进一步出示,利用Ddd_Ss构造的线粒体碱基编辑器,成功在多种细胞中实现了对14个线粒体基因组特定位点的编辑。通过引入David Liu等人之前发现的提高脱氨酶活性的突变,作者成功构建了模拟Leber遗传性视神经病变中的线粒体基因突变,发现这些突变会导致线粒体功能的减弱(图2c,d)。有意思的是,反过来将Ddd_Ss中一个位点(N1370)引入到伯克霍尔德菌的DddA(Ddd_Bc)时,成功提高了Ddd_Bc衍生的线粒体碱基编辑器的活性和序列兼容性。这些研究拓展了线粒体碱基编辑器的序列兼容性,并为未来开发新的线粒体碱基编辑器提供资源和参考。
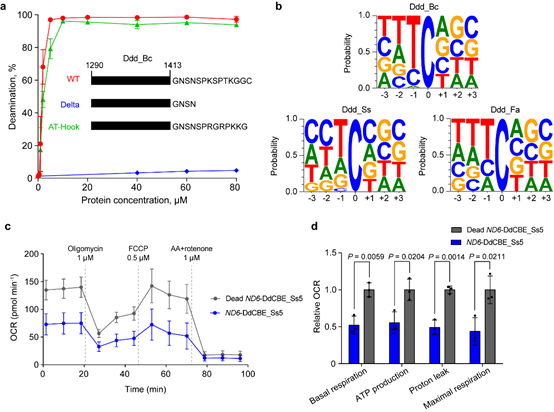
图2. DddA羧基端序列的重要性(a),三种不同来源的DddA的序列偏好(b),及经Dd_Ss衍生的线粒体碱基编辑器编辑后的细胞线粒体功能分析(c,d)。
该研究拓展了线粒体碱基编辑器的序列兼容性,但并没有解决该领域目前广泛存在的脱靶问题,未来需要更多更精巧的设计来实现高效和高特异性的编辑;在此之前,线粒体碱基编辑器的应用仍然会面临挑战。
北京大学未来技术学院博士生米黎和北大-清华生命科学联合中心博士生石铭为该论文共同第一作者。该研究得到了北京大学伊成器、高宁、魏文胜、程和平、王显花和陈良怡实验室的指导与帮助;研究由国家自然科学基金委和国家重点研发计划“干细胞研究与器官修复”专项资助,新发现的双链脱氨酶相关应用申请专利一项。
1 Mi, L. et al. DddA homolog search and engineering expand sequence compatibility of mitochondrial base editing. Nature Communications 14 (2023). https://doi.org:10.1038/s41467-023-36600-2
2 Mok, B. Y. et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature 583, 631-637 (2020). https://doi.org:10.1038/s41586-020-2477-4
3 Mok, B. Y. et al. CRISPR-free base editors with enhanced activity and expanded targeting scope in mitochondrial and nuclear DNA. Nat Biotechnol (2022). https://doi.org:10.1038/s41587-022-01256-8
