随着单细胞转录组测序(scRNA-seq)技术的成熟,来自不同生物学系统的单细胞转录组数据不断累积,为揭示细胞类型在组织稳态、发育和病理状态下的基因表达异质性提供了重要的数据基础。然而,在组织解离过程中,单细胞的空间位置信息会丢失,限制了单细胞技术在解析与空间相关的分子特征方面的应用,如空间尺度的细胞间相互作用等。近年来,新兴的空间转录组学(Spatial Transcriptomics)技术快速发展,在实现基因表达水平的测量的同时,保留了原始的空间位置信息。因此,如果能将空间转录组学数据作为参考,用于指导单细胞组学数据的空间位置重构,有望提升海量单细胞组学数据的附加价值。
2023年5月24日,北京大学未来技术学院丁晚秋、李川昀团队在 Nucleic Acids Research 杂志发表题为“ STellaris: a web server for accurate spatial mapping of single cells based on spatial transcriptomics data ” 的研究论文,开发了网页端应用STellaris,可通过整合空间转录组学和单细胞转录组学数据,实现对单细胞组学数据的空间重构。进一步,利用重构的单细胞精度的空间转录组图谱,STellaris可以提供空间范围内的细胞间相互作用图谱,例如细胞的空间邻近关系和受体-配体相互作用等。

首先,通过整合公共数据资源,作者搭建了空间转录组学数据库作为空间比对的数据参考集合,其中收录了来自人和小鼠总计101套空间转录组数据,包含了823张组织切片。特别地,他们利用Stereo-seq技术产出一套E14.5小鼠胚胎大脑冠状面的空间转录组数据,并囊括在数据库中。其次,作者设计了一套筛选体系,能够帮助用户快速定位与其输入的单细胞组学数据匹配的空间转录组切片。同时,为了消除由于单细胞组学与空间转录组学数据不完全匹配所引入的假阳性的重构结果,作者引入了系统的质控评估体系,能够过滤无法有效整合的单细胞,并对重构结果的质量给予评估。在空间重构环节,作者整合并改进了目前流行的基于配套空间转录组数据实现单细胞空间重构的算法,以确保网页服务器的通用性。最后,基于重构的单细胞精度的空间转录组图谱,STellaris进一步描绘空间尺度的细胞间相互作用图谱,能够帮助用户找到邻近的细胞类型对及潜在的受体-配体相互作用。值得一提的是,利用转录组学作为连接桥梁,STellaris能够同时实现单细胞多组学数据的空间位置重构,帮助用户探究空间尺度的基因表达调控机制。
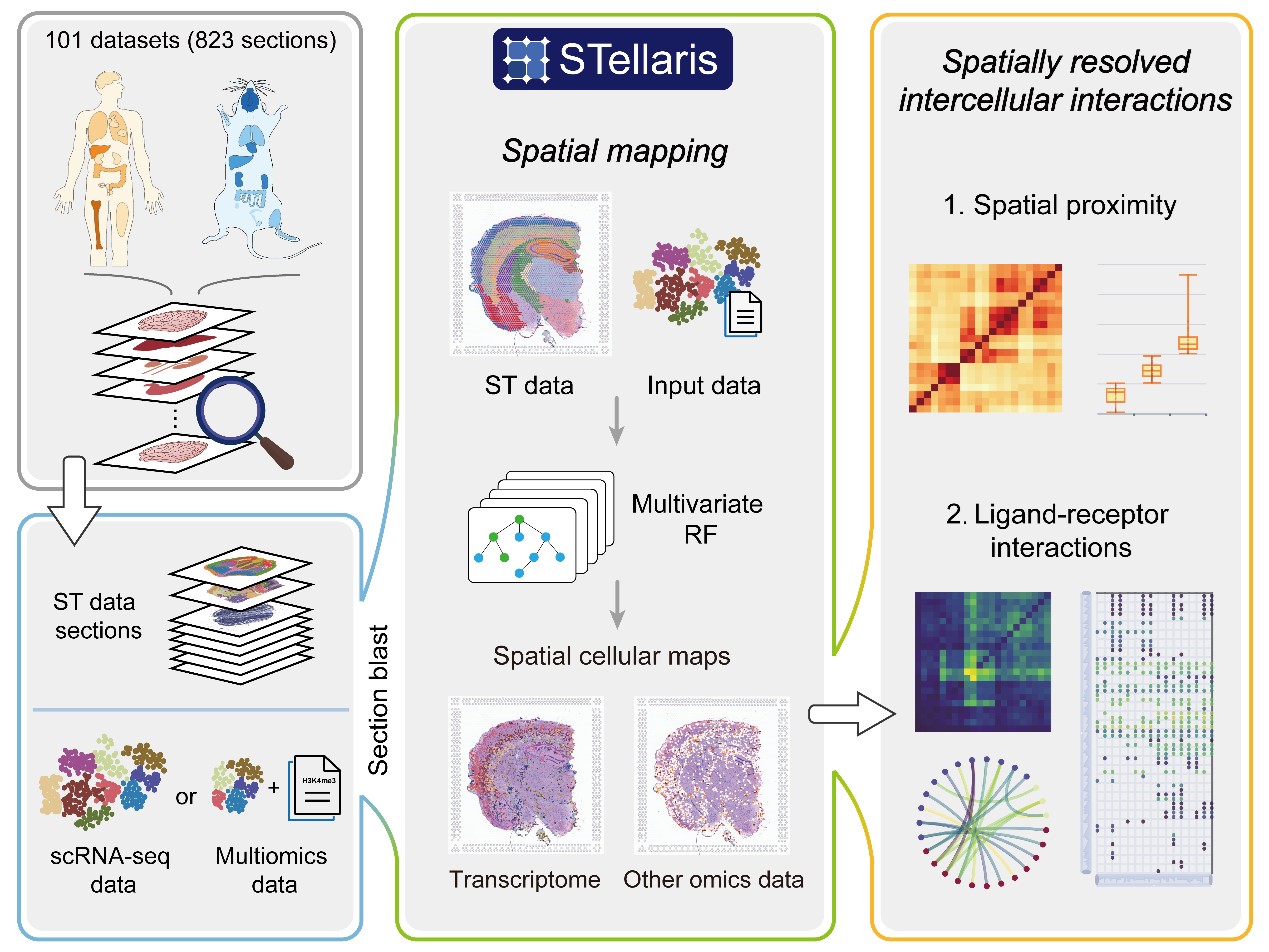
STellaris提供了用户友好的界面和丰富多样的功能,并已被应用于不同的生物学系统的案例中,包括胚胎时期小鼠大脑皮层发生过程、人类鳞状细胞癌肿瘤微环境内的细胞相互作用和小鼠大脑H3K4me3组蛋白修饰的空间特征。
北京大学未来技术学院李川昀课题组李向上、肖淳夫和齐俊添同学担任本文共同第一作者,北京大学未来技术学院丁晚秋博士、李川昀教授担任通讯作者。北京华大生命科学研究院与北京大学分子医学南京转化研究院南京脑观象台也对本研究给予了支持。该研究得到了国家重点研发计划、国家自然科学基金和北京脑科学与类脑研究中心的支持。
原文链接:
https://doi.org/10.1093/nar/gkad419
网站链接:
https://spatial.rhesusbase.com
